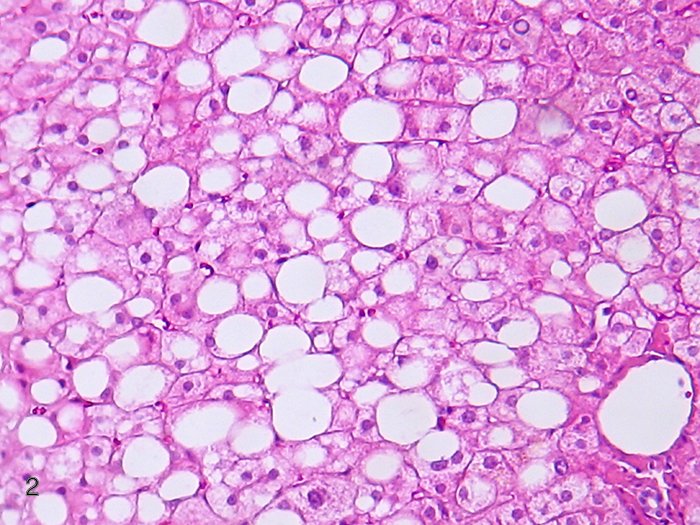
En la enfermedad del hígado graso no alcohólico (HGNA), una patología que afecta a cerca de un cuarto de la población en el mundo, la genética de los pacientes condiciona el perfil de ADN de las bacterias que colonizan el hígado o microbioma hepático, revelaron investigadores argentinos.
Los resultados son un paso importante en la comprensión de los mecanismos moleculares de una enfermedad que se caracteriza por la inflamación del hígado y puede progresar a cirrosis e insuficiencia hepática, y podrían orientar estrategias selectivas de prevención y tratamiento.
“Los hallazgos refuerzan el concepto de que los factores que llevarían al desarrollo de la HGNA son en parte heredados y en parte adquiridos”, indicó la co-líder del estudio, la doctora en Medicina y magíster en Biología Molecular Médica Silvia Sookoian, del Instituto de Investigaciones Médicas (IDIM), un Instituto de alianza estratégica UBA-CONICET.
Estudios de las comunidades bacterianas del cuerpo humano demostraron que la microbiota o comunidad de bacterias varía sistemáticamente en los hábitats del cuerpo. “Esas bacterias, dependiendo de cuál sea su especie o la presencia de su ADN en el hígado, puede desencadenar diversas reacciones inflamatorias en el tejido o a la inversa, puede condicionar propiedades beneficiosas. La composición de la microbiota tendría un rol importante en la gravedad de la HGNA”, indicó Sookoian, también investigadora principal del CONICET.
Sookoian y colegas analizaron muestras de biopsias hepáticas de 116 pacientes que tenían diferentes estadios de severidad de la HGNA; recogieron muestras de sangre para evaluar su perfil genético; hicieron análisis moleculares del microbioma tisular hepático y combinaron todos estos datos con la información clínica.
Tal como revela la revista ‘EBioMedicine’, los investigadores observaron que la combinación de ciertas bacterias o presencia de ADN bacteriano en las bacterias está muy ligadas a la predisposición genética de la enfermedad. Por ejemplo, comprobaron una asociación entre variantes genéticas y la presencia del ADN de una bacteria del género Tyzzerella que estaría muy ligada al riesgo de enfermedad cardiovascular.
“Este hallazgo permite también avanzar en el entendimiento de cómo la enfermedad grasa del hígado se conecta con otras enfermedades muy relacionadas, y hasta podríamos afirmar que todas forman parte del mismo trastorno metabólico subyacente. En realidad, la enfermedad grasa del hígado probablemente sea un componente más del llamado síndrome metabólico, la tétrada compuesta por obesidad abdominal, diabetes, hipertensión arterial y alteración de los lípidos circulantes en la sangre”, destacó Carlos J. Pirola, investigador Superior del CONICET en el IDIM.
Los científicos también encontraron que una proporción importante (alrededor del 7’4 por ciento) de la variación del perfil del microbioma bacteriano estaría explicado por la suma de 5 variantes genéticas (PNPLA3, TM6SF2, MBOAT7, HSD17B13 y FGF21) que están ligadas al riesgo de enfermedad e ingesta de azúcares. “Esto representa un dato muy importante ya que sólo caracterizamos una fracción muy pequeña de variantes genéticas”, subrayó la científica.
Actualmente, los tratamientos de la enfermedad grasa del hígado son muy limitados porque están en etapas de experimentación y porque todavía no demostraron resolver la complejidad de la enfermedad y de las lesiones que presenta el hígado enfermo, tal como la fibrosis hepática.
“Consideramos que la información revelada en nuestro trabajo, así como la línea de investigación en general nos permiten entender los mecanismos de la enfermedad. Pero tal vez lo más importante, es que esta investigación ofrece una plataforma sobre la cual evaluar futuras intervenciones terapéuticas”, indicó Sookoian.
Y agregó que “específicamente, el perfil del microbioma hepático sugiere que las opciones terapéuticas, incluida la selección de probióticos, deben definirse con precisión de acuerdo con escenarios clínicos específicos, incluidas las características del microbioma del paciente y de la carga genética del mismo”.
Del estudio también participaron Carlos Jose Pirola, primer autor del estudio e investigador del CONICET en el IDIM; Adrian Salatino y María Florencia Quintanilla también en el IDIM; Martin Garaycoechea, en el Hospital El Cruce; y Gustavo Castaño, del Hospital Zubizarreta, en Buenos Aires.